科研人员研发出一种不依赖脱氨酶的新型碱基编辑器
2023年5月16日,中国科学出版社出版的综合性学术期刊《国家科学评论》(NationalScience Review, NSR)发表了题为“Programmable deaminase-free baseeditors for G-to-Y conversion by engineered glycosylase”的研究论文,公布了一种前所未有的鸟嘌呤(G)碱基编辑器。辉大基因创始人&首席科学顾问、中国科学院脑科学与智能技术卓越创新中心、上海脑科学与类脑研究中心杨辉博士,辉大基因创新研究院童华威博士为该论文的通讯作者。

碱基编辑器(Base editor,BE)是实现特定单碱基编辑和核苷酸转变的重要工具,其极大地促进了基础研究、基因治疗、动植物育种改良等领域的进展。目前主要的DNA碱基编辑器是将可编程DNA结合蛋白(例如Cas9,Cas12或TALE蛋白变体)与腺嘌呤(A;adenine)脱氨酶或胞嘧啶(C;cytosine)脱氨酶整合在一起而构建的,分别称为ABE(Adeninebase editor,腺嘌呤碱基编辑器)和CBE(Cytosine base editor,胞嘧啶碱基编辑器),以此实现A-to-G和C-to-T这样的嘌呤碱基到嘌呤碱基或嘧啶碱基到嘧啶碱基之间的转变。在此基础上,研究人员还开发了可以实现更多样编辑的碱基编辑器。比如,在CBE的基础上开发了能实现C-to-G这样的嘧啶碱基到嘌呤碱基之间转变的CGBE(C-to-G base editor)。最近,在ABE的基础上开发了能够实现A-to-T和/或A-to-C这样的嘌呤碱基到嘧啶碱基之间的转变的新型DNA碱基编辑器AYBE(adeninetransversion base editor)。
然而,目前还没有碱基编辑器可以实现对鸟嘌呤(G)的直接编辑。虽然非编辑链上的G可以通过对编辑链上的C进行编辑而间接地被编辑,但受限于原型间隔子邻近基序(PAM)约束、编辑窗口内的编辑效率等因素的影响,很多情况下仍需要对G进行直接编辑。另外,受限于现有脱氨酶能力,DNA中的G无法通过脱氨基反应后DNA修复机制转化为其他类型的碱基,使得基于脱氨基化反应来实现单碱基编辑的技术路线无法应用于对G的直接编辑。
研发团队巧妙地跳出了基于脱氨基化反应来实现单碱基编辑的思维惯性,利用糖基化酶,成功开发了实现高效鸟嘌呤单碱基编辑的、不依赖于脱氨基化反应的新型DNA碱基编辑器gGBE(glycosylase-basedGuanineBaseEditor)。该研究提出的基于工程化糖基化酶开发碱基编辑器的新策略对进一步丰富碱基编辑工具包、建立基础研究领域疾病模型、开发基因疗法等都有着非常重要的意义。
研究团队前期通过蛋白质工程化,获得了高效切除碱基次黄嘌呤(Hx)的MPG变体,鉴于Hx在结构上与G或A的相似性,研究人员检测了多种MPG变体切除G或A的能力。为了便于评估G或A切除事件的发生以及相应的碱基编辑效率,研究人员设计了基于内含子剪接的可激活荧光报告系统。在该荧光报告系统中,只有发生了G或A的切除且实现了相应碱基的转变,才能纠正内含子剪接信号,使剪接过程正确发生,进而激活绿色荧光蛋白(EGFP)的正常表达,以便结合流式细胞术检测碱基编辑后产生的绿色荧光信号。
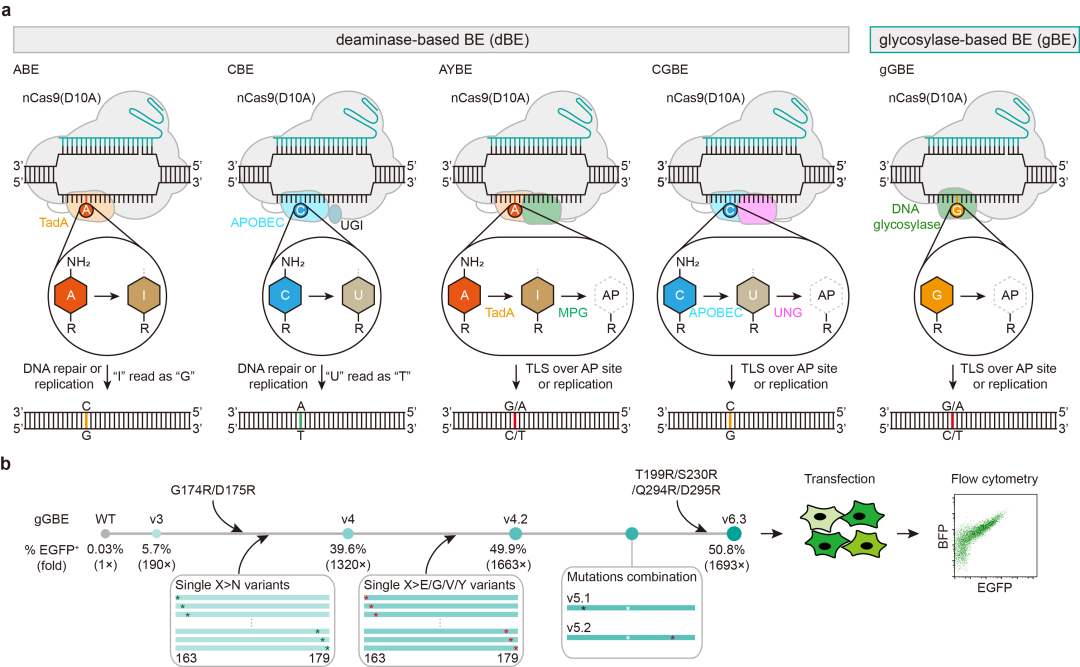
图1:基于脱氨酶(dBE)与基于糖基化酶(gBE)的碱基编辑器的比较,以及gGBE的蛋白质工程化设计和筛选
研究人员将不同版本的MPG变体融合在nCas9的C末端,构建了多个不依赖脱氨酶的、基于糖基化酶的碱基编辑器(deaminase-free, glycosylase-based base editor;gBE)。分别利用A-to-T荧光报告系统和G-to-T荧光报告系统进行评估,研究人员发现带有MPGv0.2至MPGv3变体的gBE都能检测到G-to-T编辑,而没有检测到A-to-T编辑。带有MPGv3的gBE具有最高G-to-T编辑,命名为gGBEv3。为了提升gGBE的编辑效率,研究人员结合蛋白结构分析,对gGBE中的MPG组分进行了理性设计,制定了多种工程化改造策略,构建了一系列突变体库,并利用上述G-to-T荧光报告系统在哺乳动物细胞中开展了多轮的优化筛选,获得了鸟嘌呤编辑效率最高的gGBEv6.3,其鸟嘌呤编辑效率(50.8%)是带有野生型MPG的gGBEv0.1(0.03%)的约1693倍,提升极为显著(图1)。多个gGBE版本的鸟嘌呤编辑效率也在两个基因组內源位点上得到了验证(鸟嘌呤的总体编辑效率分别从6.4%和7.5%提高到了78.5%和80.3%),而且表现出了较低的indels频率。
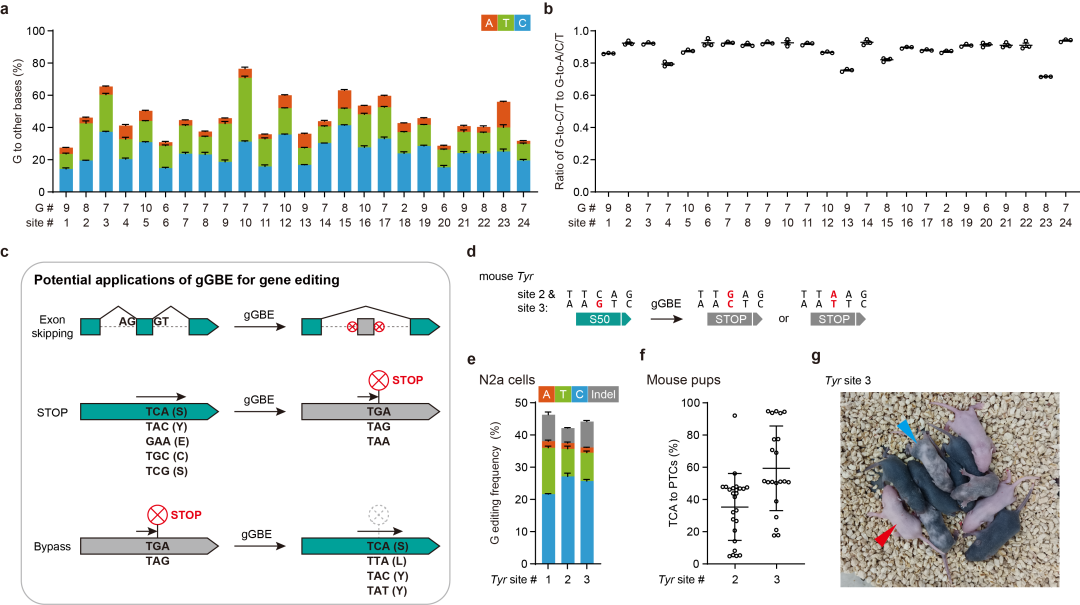
图2:gGBEv6.3在內源位点上的编辑效率评估及相关应用示例
研究人员还在人类基因组上的24个靶点对gGBEv6.3作了综合评估,发现其可实现高达81.2%的鸟嘌呤编辑效率,产物主要是G-to-C和G-to-T编辑,总体G-to-Y(Y = C or T)编辑占比可达95%(图2)。通过sgRNA依赖性和sgRNA非依赖性脱靶分析,研究人员发现gGBEv6.3的DNA脱靶编辑水平较低。研究人员还探究了gGBEv6.3在剪接位点编辑、提前终止密码子(prematuretermination codon,PTC)引入等方面的应用潜力。研究人员发现gGBEv6.3可以有效编辑DMD基因第45号外显子的剪接受体位点(总体编辑效率达30.3%)。进一步的研究发现,gGBEv6.3在小鼠细胞系及胚胎中均表现出高效的编辑效率。在新生小鼠中检测到PTC引入效率平均达59.4%,最高可达94.87%,并且只产生很少的indels。超过57%的F0代小鼠表现出白化或嵌合表型(图2)。
在所有人类致病性SNP(共60372个)中,约有5%的T-to-GSNP和10%的C-to-G SNP。对于T-to-G SNP,目前没有任何碱基编辑器可以有效地直接实现G到T纠正或进行反义链中的C到A转化来间接实现G到T纠正。对于C-to-G SNP,尽管CGBE理论上可以通过进行反义链中的C到G转化来间接实现G到C纠正,但由于PAM的严格约束和狭窄的编辑窗口,通常难以设计有效的sgRNA来实现,而利用gGBE,则可以直接实现G到C纠正。因此,gGBE的开发极大拓宽了碱基编辑器的适用范围。