上海科学家开发出通用且高效的哺乳动物基因敲入策略
2025年7月15日,中国科学院脑科学与智能技术卓越创新中心、上海脑科学与类脑研究中心的刘真研究组开发出通用且高效的哺乳动物基因敲入策略。研究成果以“Refined DNA repair manipulation enables a universal knock-in strategy in mouse embryos”发表在《Nature Communications》期刊上。
本研究聚焦于哺乳动物胚胎基因敲入效率低,编辑效果存在位点偏好等技术瓶颈,开发出一种适用于不同基因位点、敲入效率最高可达90%的CRISPR通用敲入策略——ChemiCATI(图1),实现了高效、可预测的哺乳动物胚胎基因组精准编辑,为动物模型的建立和胚胎发育研究提供了关键技术支撑。
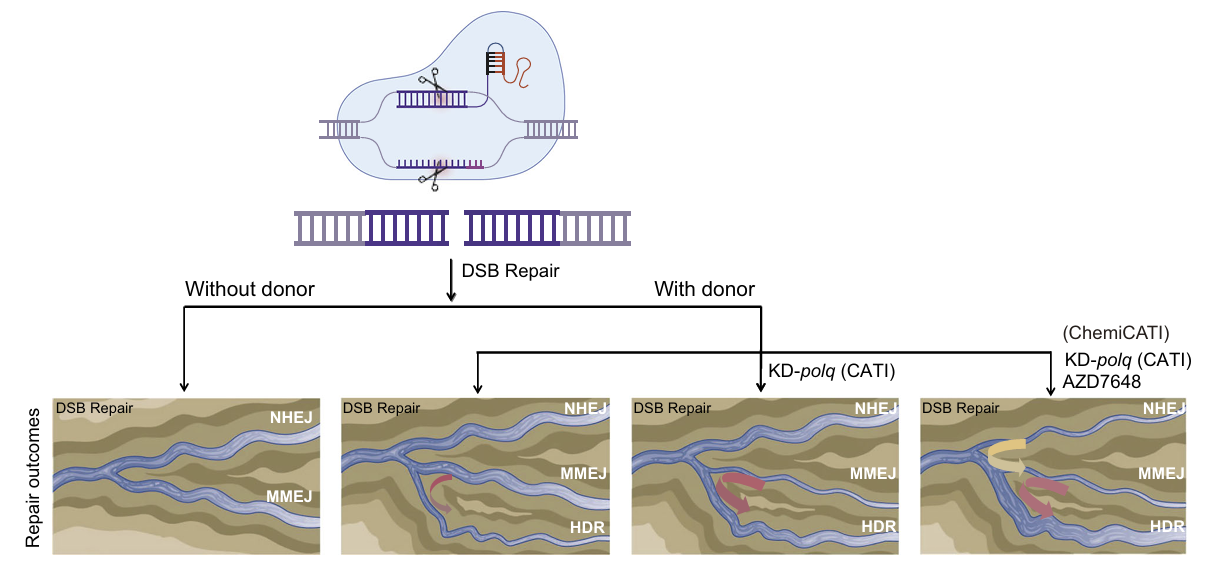
图 1 ChemiCATI策略的原理
在哺乳动物,尤其是胚胎阶段,实现精准基因敲入仍面临诸多技术挑战。CRISPR/Cas9系统作为当前基因组精准编辑的核心工具,其诱导的DNA双链断裂(DSBs)可通过多种通路进行修复,包括非同源末端连接(NHEJ)、微同源末端连接(MMEJ)以及同源重组修复(HDR),其中HDR是实现外源基因精准整合的关键通路。但HDR在哺乳动物胚胎中发生概率低,严重制约了高效敲入策略的建立。
针对上述难题,刘真研究组在前期研究中发现,哺乳动物胚胎中CRISPR引发的DSB主要依赖MMEJ通路进行修复。在此基础上,团队开发了CATI(CasRx-Assisted Targeted Integration)策略,利用CRISPR/Cas13d (CasRx)系统对MMEJ关键因子Polq进行敲低,显著提高了基因敲入效率,并有效减少大片段DNA缺失(Chen et al., Genome Biology, 2023),为建立高效率、低脱靶的敲入策略奠定了基础。尽管CATI策略在多个物种中展现出良好通用性,但研究团队发现,敲入效率在很大程度上仍取决于所选用的单导向(sgRNA)的修复偏好。即使位于同一目标区域,“切割效率”相同的sgRNA之间的敲入效率差异依然显著(图2)。
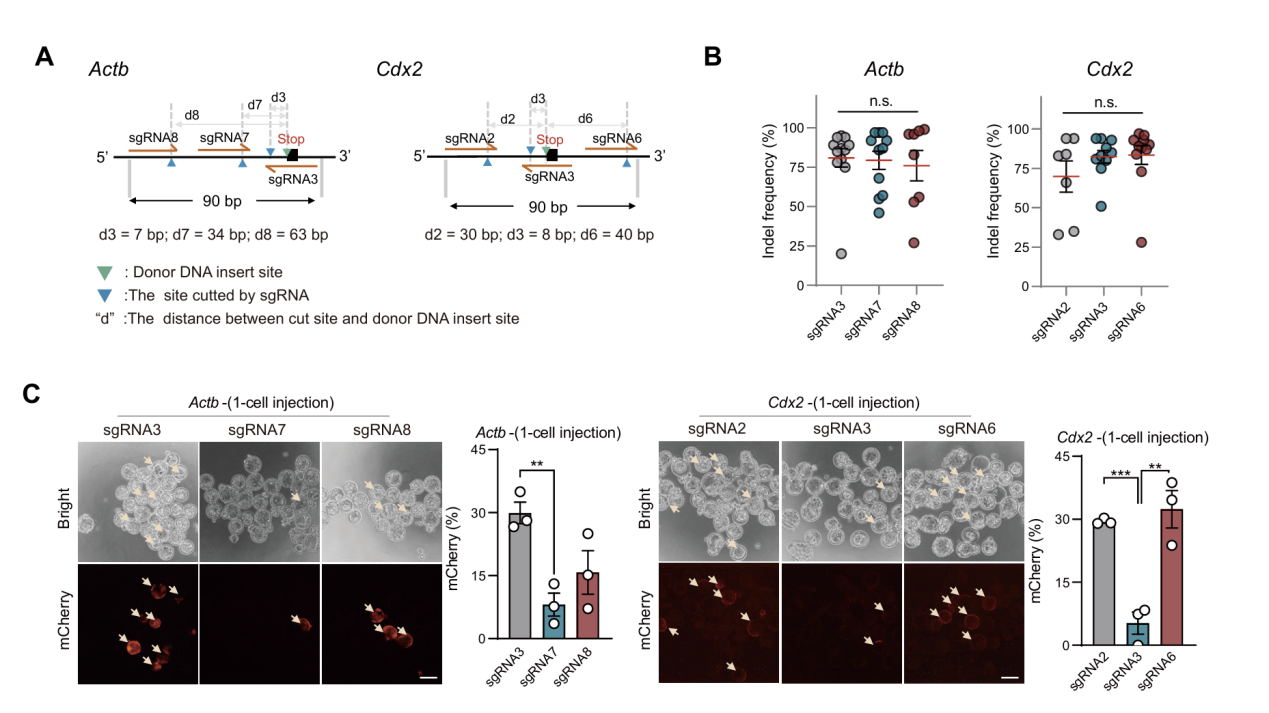
图 2 相同切割效率的sgRNA在邻近位点表现出不同的基因敲入效率
为了深入研究这一现象,研究团队系统分析了30余个基因位点、200余条sgRNA的切割修复序列特征,将sgRNA归类为MMEJ偏好型(MMEJ-biased)、NHEJ偏好型(NHEJ-biased)和平衡型(NHEJ/MMEJ-balanced)。结果表明,具有MMEJ偏好的sgRNA普遍具有更高的敲入效率,而偏向NHEJ的sgRNA敲入效率显著下降。CATI策略虽能显著提升MMEJ-biased sgRNA的敲入效果,但对NHEJ-biased sgRNA作用有限(图3)。
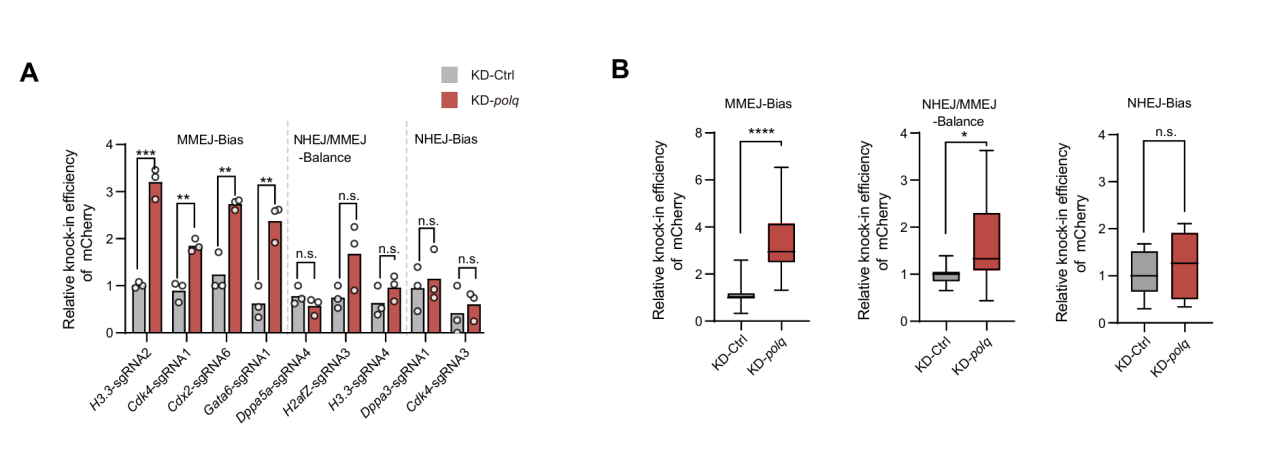
图 3 CATI策略不能有效提升NHEJ-Biased sgRNA基因敲入效率
为突破这一限制,研究团队进一步开展小分子化合物筛选,发现DNA依赖性蛋白激酶催化亚基(DNA-PKcs)抑制剂AZD7648可显著抑制NHEJ修复通路,并将DNA断裂的修复倾向由NHEJ偏向转为MMEJ,从而“重塑”sgRNA的修复途径偏好。基于此,研究团队将AZD7648与CATI策略联合应用后,提出并建立了一种适用于不同sgRNA类型和多种基因位点的通用高效HDR提升策略——ChemiCATI。在覆盖20余个基因、30多条sgRNA实验证明中,ChemiCATI策略在不同sgRNA修复偏好的背景下均能显著提升基因敲入效率,无论是单链DNA(ssDNA)还是双链DNA(dsDNA)供体模板,均表现出优越的适应性与稳定性,最高敲入效率可达90%。该策略不仅克服了传统敲入方法对“高效sgRNA”的依赖,还极大拓宽了基因编辑在临床疾病模型构建与治疗中的应用空间,尤其为sgRNA选择受限的致病突变位点提供了切实可行的技术解决方案(图 4)。
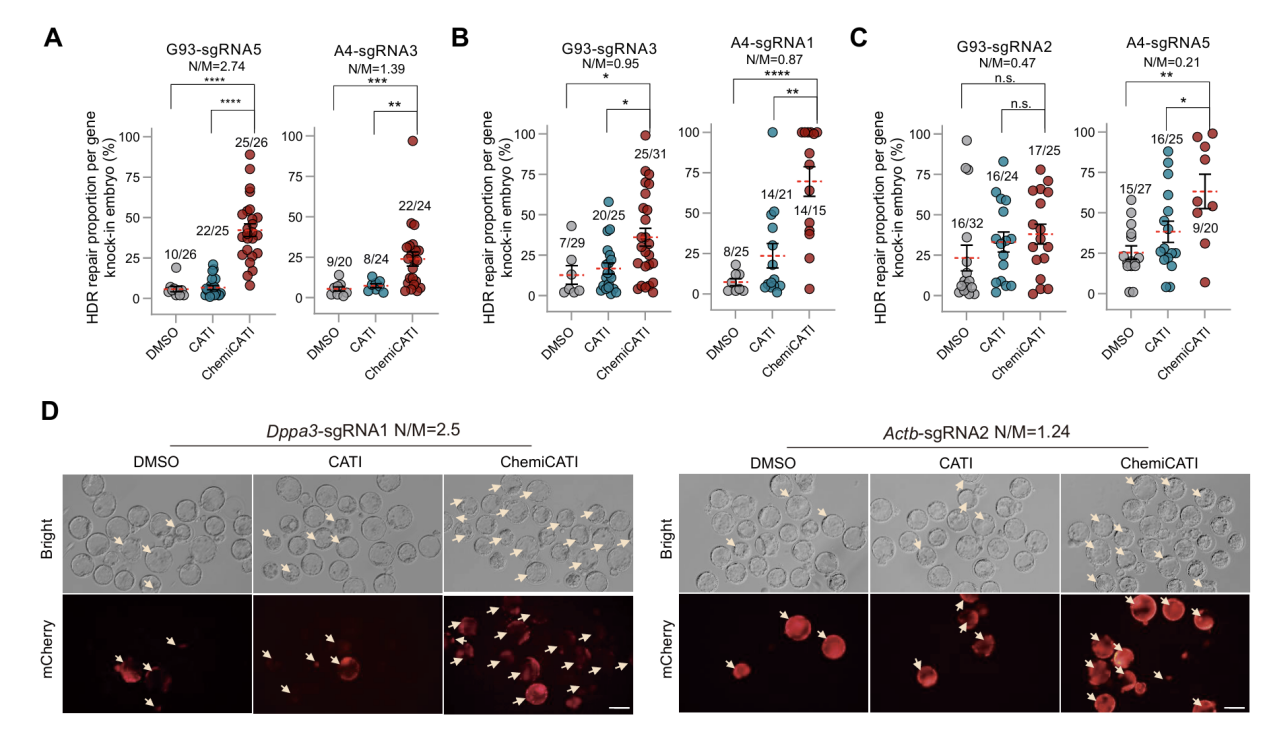
图4 ChemiCATI策略在多种sgRNA背景下显著提高ssDNA和dsDNA供体的基因敲入效率
刘真研究员、陆宗阳博士是本研究的共同通讯作者。研究得到国家重点研发计划、上海市市级重大科技专项等项目的资助。